B3 - Zusammenhang zwischen Darmmikrobiom-assoziierter bakterieller Translokation und genetischer Regulation der Körperfettverteilung
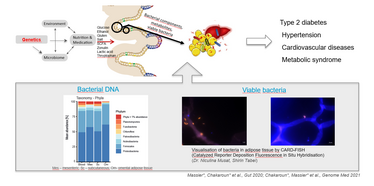
Es wird getestet, ob die bakterielle Präsenz und Zusammensetzung im Fettgewebe die erhöhte intestinale Permeabilität reflektiert, und ob dies zum Entzündungsstatus und zum dysfunktionalem Fettgewebe führt. Diese Ereignisse interagieren mit dem individuellen genetischen Hintergrund und gehen mit den metabolischen Konsequenzen der Adipositas und der Fettverteilung einher.
Studien in Tiermodellen unter fettreicher Ernährung legen nahe, dass Adipositaserkrankungen durch Bakterien-induzierte Darmentzündung, Beeinträchtigung der intestinalen Barriereintegrität und über die Translokation lebender Bakterien vom Darm in das mesenteriale Fettgewebe entstehen könnten. Im mesenterialen, viszeralen, epiploischen und subkutanen Fettgewebe von 100 Personen mit und von 25 Kontrollen ohne Adipositas wird die Präsenz und die Art der Bakterien mittels 16S rRNA Gensequenzierung ermittelt. Zur Verifizierung vitaler Bakterien werden CARD-FISH (catalyzed reporter deposition - fluorescence in situ hybridization) und Rasterelektronenmikroskopie verwendet. Ein Lactulose-Mannitol-Test wird durchgeführt, um die Darmpermeabilität als möglichen Auslöser der bakteriellen Translokation zu messen, und Biomarker der intestinalen Permeabilität (z.B. Calprotectin) werden ermittelt. Um zu testen, ob der Zusammenhang zwischen der Genetik und den metabolischen Konsequenzen der nachteiligen Fettverteilung durch Bakterien im Fettgewebe vermittelt wird, werden Genotypen und Expression der Fettverteilungsgene in Bezug auf den metabolischen und inflammatorischen Status der Probanden als auch auf die individuelle bakterielle Zusammensetzung im Fettgewebe analysiert. Schließlich werden die möglichen Auswirkungen der Bakterien im Fettgewebe untersucht, indem die humanen Adipozyten in vitro mit bakterieller DNA und mit den Spezies behandelt werden, die mit dem inflammatorischen und dem gestörten Stoffwechselprofil der untersuchten Probanden zusammenhängen.
Abbildung 1: Hypothese - die Bakterienbelastung und -zusammensetzung im Fettgewebe spiegeln eine erhöhte intestinale Permeabilität wider, die zu einem entzündlichen Zustand und einer Dysfunktion des Fettgewebes führt
Würfel M, Breitfeld J, Gebhard C, Scholz M, Baber R, Riedel-Heller SG, Blüher M, Stumvoll M, Kovacs P, Tönjes A. Interplay between adipose tissue secreted proteins, eating behavior and obesity. Eur J Nutr. 2021 Oct 12.
Krieg L, Didt K, Karkossa I, Bernhart SH, Kehr S, Subramanian N, Lindhorst A, Schaudinn A, Tabei S, Keller M, Stumvoll M, Dietrich A, von Bergen M, Stadler PF, Laurencikiene J, Krüger M, Blüher M, Gericke M, Schubert K, Kovacs P, Chakaroun R, Massier L. Gut. 2021 Oct 1:gutjnl-2021-324603.
Chakaroun RM, Massier L, Heintz-Buschart A, Said N, Fallmann J, Crane A, Schütz T, Dietrich A, Blüher M, Stumvoll M, Musat N, Kovacs P. Circulating bacterial signature is linked to metabolic disease and shifts with metabolic alleviation after bariatric surgery. Genome Med. 2021 Jun 22;13(1):105.
Chen J, … Kiess W, … Körner A, Kovacs P,…, Stumvoll M, …Tönjes A,... , Barroso I. The trans-ancestral genomic architecture of glycemic traits. Nat Genet. 2021 Jun;53(6):840-860.
Sun C, Kovacs P, Guiu-Jurado E. Genetics of Body Fat Distribution: Comparative Analyses in Populations with European, Asian and African Ancestries. Genes (Basel). 2021 May 29;12(6):841.
Yaskolka Meir A, Keller M, Müller L, Bernhart SH, Tsaban G, Zelicha H, Rinott E, Kaplan A, Gepner Y, Shelef I, Schwarzfuchs D,[nbsp]Ceglarek U,[nbsp]Stadler P, Blüher M, Stumvoll M, Kovacs P, Shai I. Effects of lifestyle interventions on epigenetic signatures of liver fat: CENTRAL randomized controlled trial. Liver Int. 2021 May 3.
Epperlein S, Gebhardt C, Rohde K, Chakaroun R, Patt M, Schamarek I, Kralisch S, Heiker JT, Scholz M, Stumvoll M, Kovacs P, Breitfeld J, Tönjes A. The Effect of FGF21 and Its Genetic Variants on Food and Drug Cravings, Adipokines and Metabolic Traits. Biomedicines. 2021 Mar 29;9(4):345.
Massier L, Blüher M, Kovacs P, Chakaroun RM. Impaired Intestinal Barrier and Tissue Bacteria: Pathomechanisms for Metabolic Diseases. Front Endocrinol (Lausanne). 2021 Mar 9;12:616506.
Meir A Y, Keller Maria, Bernhart S H, Rinott E, Tsaban G, Zelicha H, Kaplan A, Schwarzfuchs D, Shelef I, Gepner Y, Li J, Lin Y, Blüher M, Ceglarek U, Stumvoll M, Stadler P F, Stampfer M J, Kovacs P, Liang L, Shai I. Lifestyle weight‑loss intervention may attenuate methylation aging: the CENTRAL MRI randomized controlled trial. Clin Epigenetics. 2021 Mar 4;13(1):48.
Keller M, Yaskolka Meir A, Bernhart SH, Gepner Y, Shelef I, Schwarzfuchs D, Tsaban G, Zelicha H, Hopp L, Müller L, Rohde K, Böttcher Y, Stadler PF, Stumvoll M, Blüher M, Kovacs P, Shai I. DNA methylation signature in blood mirrors successful weight-loss during lifestyle interventions: the CENTRAL trial. Genome Med. 2020 Nov 16;12(1):97.
Sun C, Kovacs P, Guiu-Jurado E. Genetics of Obesity in East Asians. Front Genet. 2020 Oct 20;11:575049.
Landgraf K, Klöting N, Gericke M, Maixner N, Guiu-Jurado E, Scholz M, Witte AV, Beyer F, Schwartze JT, Lacher M, Villringer A, Kovacs P, Rudich A, Blüher M, Kiess W, Körner A. The Obesity-Susceptibility Gene TMEM18 Promotes Adipogenesis through Activation of PPARG. Cell Rep. 2020 Oct 20;33(3):108295.
Massier L, Chakaroun R, Kovacs P, Heiker JT. Blurring the picture in leaky gut research: how shortcomings of zonulin as a biomarker mislead the field of intestinal permeability. Gut . 2020 Oct 9;gutjnl-2020-323026.
Schleinitz D, Krause K, Wohland T, Gebhardt C, Linder N, Stumvoll M, Blüher M, Bechmann I, Kovacs P, Gericke M, Tönjes A. Identification of distinct transcriptome signatures of human adipose tissue from fifteen depots. Eur J Hum Genet. 2020 Jul 13.
Kunath A, Heiker JT, Kern M, Kosacka J, Flehmig G, Stumvoll M, Kovacs P, Blüher M, Klöting N. Nicotinamide Nucleotide Transhydrogenase (Nnt) Is Related to Obesity in Mice. Horm Metab Res . 2020 Jul 6.
Keller M, Gebhardt C, Huth S, Schleinitz D, Heyne H, Scholz M, Stumvoll M, Böttcher Y, Tönjes A, Kovacs P. Genetically programmed changes in transcription of the novel progranulin regulator. J Mol Med (Berl) . 2020 Jul 3.
Massier L, Chakaroun R, Tabei S, Crane A, Didt KD, Fallmann J, von Bergen M, Haange SB, Heyne H, Stumvoll M, Gericke M, Dietrich A, Blüher M, Musat N, Kovacs P. Adipose tissue derived bacteria are associated with inflammation in obesity and type 2 diabetes. Gut. 2020 Apr 21. pii: gutjnl-2019-320118.
Chakaroun RM, Massier L, Kovacs P. Gut Microbiome, Intestinal Permeability, and Tissue Bacteria in Metabolic Disease: Perpetrators or Bystanders? Nutrients. 2020 Apr 14;12(4).
Breitfeld J, Kehr S, Müller L, Stadler PF, Böttcher Y, Blüher M, Stumvoll M, Kovacs P. Developmentally Driven Changes in Adipogenesis in Different Fat Depots Are Related to Obesity. Front Endocrinol (Lausanne). 2020 Mar 26;11:138.
Jacobi T, Massier L, Klöting N, Horn K, Schuch A, Ahnert P, Engel C, Löffler M, Burkhardt R, Thiery J, Tönjes A, Stumvoll M, Blüher M, Doxiadis I, Scholz M, Kovacs P. HLA Class II Allele Analyses Implicate Common Genetic Components in Type 1 and Non–Insulin-Treated Type 2 Diabetes. J Clin Endocrinol Metab. 2020 Mar 1;105(3).
Teumer A, [...], Körner A, Kovacs P, [...], Stumvoll M, [...], Köttgen A. Genome-wide association meta-analyses and fine-mapping elucidate pathways influencing albuminuria. Nat Commun. 2019 Sep 11;10(1):4130.
Weingarten MFJ, Scholz M, Wohland T, Horn K, Stumvoll M, Kovacs P, Tönjes A. Circulating oxytocin is genetically determined and associated with obesity and impaired glucose tolerance. J Clin Endocrinol Metab. 2019 Jul 30. pii: jc.2019-00643
Heinitz S, Gebhardt C, Piaggi P, Krüger J, Heyne H, Weiner J, Heiker JT, Stumvoll M, Blüher M, Baier L, Rudich A, Kovacs P, Tönjes A. Atg7-knockdown reduces chemerin secretion in murine adipocytes. J Clin Endocrinol Metab. 2019 Jun 21. pii: jc.2018-01980.
Wuttke M,[...], Kiess W, Kleber M, Koenig W, Kooner JS, Körner A, Kovacs P,[...], Stumvoll M., Pattaro C. A catalog of genetic loci associated with kidney function from analyses of a million individuals. Nat Genet. 2019 Jun;51(6):957-972.
Warrington NM, [...], Kovacs P, [...], Tönjes A, Stumvoll M, Michaelsen KF, Eloranta AM, Lakka TA, van Duijn CM, Kiess W, Körner A, [...], Freathy RM. Maternal and fetal genetic effects on birth weight and their relevance to cardio-metabolic risk factors. Nat Genet. 2019 May;51(5):804-814.
Rohde K, Keller M, la Cour Poulsen L, Rønningen T, Stumvoll M, Tönjes A, Kovacs P, Horstmann A, Villringer A, Blüher M, Böttcher Y. (Epi)genetic regulation of CRTC1 in human eating behaviour and fat Distribution. EBioMedicine. 2019 May 29. pii: S2352-3964(19)30358-5.
Breitfeld J, Wiele N, Gutsmann B, Stumvoll M, Blüher M, Scholz M, Kovacs P, Tönjes A. Circulating Adipokine VASPIN Is Associated with Serum Lipid Profiles in Humans. Lipids. 2019 Mar 18.
Bartels MS, Scheffler L, Chakaroun R, Dietrich A, Blüher M, Stumvoll M, Tönjes A, Breitfeld J, Kovacs P. Increased circulating cell-free DNA in insulin resistance.Diabetes Metab. 2018 Jun 20.
Goldstein N, Haim Y, Mattar P, Hadadi-Bechor S, Maixner N, Kovacs P, Blüher M, Rudich A. Leptin stimulates autophagy/lysosome-related degradation of long-lived proteins in adipocytes. Adipocyte. 2019 Jan 24.
Rohde K, Keller M, la Cour Poulsen L, Blüher M, Kovacs P, Böttcher Y. Genetics and epigenetics in obesity. Metabolism. 2018 Nov 3. pii: S0026-0495(18)30225-7.
Keller M, Klös M, Rohde K, Krüger J, Kurze T, Dietrich A, Schön MR, Gärtner D, Lohmann T, Dreßler M, Stumvoll M, Blüher M, Kovacs P, Böttcher Y. DNA methylation of SSPN is linked to adipose tissue distribution and glucose metabolism. FASEB J. 2018 Jun 22:fj201800528R.
Krüger J, Berger C, Weidle K, Schleinitz D, Tönjes A, Stumvoll M, Blüher M, Kovacs P, Klöting N. Metabolic effects of genetic variation in the human REPIN1 gene. Circ Genom Precis Med. 2018 May;11(5):e001992.
Huhn S, Beyer F, Zhang R, Lampe L, Grothe J, Kratzsch J, Willenberg A, Breitfeld J, Kovacs P, Stumvoll M, Trampel R, Bazin PL, Villringer A, Witte AV. Effects of resveratrol on glucose, memory and the hippocampus - a randomized controlled trial. Neuroimage. 2018 Mar 13;174:177-190.
Scheffler L, Crane A, Heyne H, Tönjes A, Schleinitz D, Ihling HC, Stumvoll M, Freire R, Fiorentino M, Fasano A, Kovacs P and Heiker JT. Widely Used commercial elisa. Does not Detect Precursor of haptoglobin2, but recognizes Properdin as a Potential second Member of the Zonulin Family. Front Endocrinol (Lausanne). 2018 Feb 5;9:22
Tönjes A, Scholz M, Krüger J, Krause K, Schleinitz D, Kirsten H, Gebhardt C, Marzi C, Grallert H, Ladenvall C, Heyne H, Laurila E, Kriebel J, Meisinger C, Rathmann W, Gieger C, Groop L, Prokopenko I, Isomaa B, Beutner F, Kratzsch J, Fischer-Rosinsky A, Pfeiffer A, Krohn K, Spranger J, Thiery J, Blüher M, Stumvoll M, Kovacs P. Genome-wide meta-analysis identifies novel determinants of circulating serum progranulin. Hum Mol Genet. 2018 Feb 1;27(3):546-558.
Gross A, Tönjes A, Scholz M. On the impact of relatedness on SNP association analysis. BMC Genet. 2017;18:104.
Rohde K, Klös M, Hopp L, Liu X, Keller M, Stumvoll M, Dietrich A, Schön MR, Gärtner D, Lohmann T, Dreßler M, Kovacs P, Binder H, Blüher M, Böttcher Y. IRS1 DNA promoter methylation and expression in human adipose tissue are related to fat distribution and metabolic traits. Sci Rep. 2017;7:12369.
Rosendahl J, Kirsten H, Hegyi E, Kovacs P, Weiss FU, Laumen H, Lichtner P, Ruffert C, Chen JM, Masson E, Beer S, Zimmer C, Seltsam K, Algül H, Bühler F, Bruno MJ, Bugert P, Burkhardt R, Cavestro GM, Cichoz-Lach H, Farré A, Frank J, Gambaro G, Gimpfl S, Grallert H, Griesmann H, Grützmann R, Hellerbrand C, Hegyi P, Hollenbach M, Iordache S, Jurkowska G, Keim V, Kiefer F, Krug S, Landt O, Leo MD, Lerch MM, Lévy P, Löffler M, Löhr M, Ludwig M, Macek M, Malats N, Malecka-Panas E, Malerba G, Mann K, Mayerle J, Mohr S, Te Morsche RHM, Motyka M, Mueller S, Müller T, Nöthen MM, Pedrazzoli S, Pereira SP, Peters A, Pfützer R, Real FX, Rebours V, Ridinger M, Rietschel M, Rösmann E, Saftoiu A, Schneider A, Schulz HU, Soranzo N, Soyka M, Simon P, Skipworth J, Stickel F, Strauch K, Stumvoll M, Testoni PA, Tönjes A, Werner L, Werner J, Wodarz N, Ziegler M, Masamune A, Mössner J, Férec C, Michl P, P H Drenth J, Witt H, Scholz M, Sahin-Tóth M; all members of the PanEuropean Working group on ACP. Genome-wide association study identifies inversion in the CTRB1-CTRB2 locus to modify risk for alcoholic and non-alcoholic chronic pancreatitis. Gut. 2017; Epub ahead of print.
Weiner J, Rohde K, Krause K, Zieger K, Klöting N, Kralisch S, Kovacs P, Stumvoll M, Blüher M, Böttcher Y, Heiker JT. Brown adipose tissue (BAT) specific vaspin expression is increased after obesogenic diets and cold exposure and linked to acute changes in DNA-methylation. Mol Metab. 2017;6:482-93.
Keller M, Hopp L, Liu X, Wohland T, Rohde K, Cancello R, Klös M, Bacos K, Kern M, Eichelmann F, Dietrich A, Schön MR, Gärtner D, Lohmann T, Dreßler M, Stumvoll M, Kovacs P, DiBlasio AM, Ling C, Binder H, Blüher M, Böttcher Y. Genome-wide DNA promoter methylation and transcriptome analysis in human adipose tissue unravels novel candidate genes for obesity. Mol Metab. 2016;6:86-100
Ried JS, Jeff M J, Chu AY, Bragg-Gresham JL, van Dongen J, Huffman JE, Ahluwalia TS, Cadby G, Eklund N, Eriksson J, Esko T, Feitosa MF, Goel A, Gorski M, Hayward C, Heard-Costa NL, Jackson AU, Jokinen E, Kanoni S, Kristiansson K, Kutalik Z, Lahti J, Luan J, Mägi R, Mahajan A, Mangino M, Medina-Gomez C, Monda KL, Nolte IM, Pérusse L, Prokopenko I, Qi L, Rose LM, Salvi E, Smith MT, Snieder H, Stančáková A, Ju Sung Y, Tachmazidou I, Teumer A, Thorleifsson G, van der Harst P, Walker RW, Wang SR, Wild SH, Willems SM, Wong A, Zhang W, Albrecht E, Couto Alves A, Bakker SJ, Barlassina C, Bartz TM, Beilby J, Bellis C, Bergman RN, Bergmann S, Blangero J, Blüher M, Boerwinkle E, Bonnycastle LL, Bornstein SR, Bruinenberg M, Campbell H, Chen YI, Chiang CW, Chines PS, Collins FS, Cucca F, Cupples LA, D'Avila F, de Geus EJ, Dedoussis G, Dimitriou M, Döring A, Eriksson JG, Farmaki AE, Farrall M, Ferreira T, Fischer K, Forouhi NG, Friedrich N, Gjesing AP, Glorioso N, Graff M, Grallert H, Grarup N, Gräßler J, Grewal J, Hamsten A, Harder MN, Hartman CA, Hassinen M, Hastie N, Hattersley AT, Havulinna AS, Heliövaara M, Hillege H, Hofman A, Holmen O, Homuth G, Hottenga JJ, Hui J, Husemoen LL, Hysi PG, Isaacs A, Ittermann T, Jalilzadeh S, James AL, Jørgensen T, Jousilahti P, Jula A, Marie Justesen J, Justice AE, Kähönen M, Karaleftheri M, Tee Khaw K, Keinanen-Kiukaanniemi SM, Kinnunen L, Knekt PB, Koistinen HA, Kolcic I, Kooner IK, Koskinen S, Kovacs P, Kyriakou T, Laitinen T, Langenberg C, Lewin AM, Lichtner P, Lindgren CM, Lindström J, Linneberg A, Lorbeer R, Lorentzon M, Luben R, Lyssenko V, Männistö S, Manunta P, Leach IM, McArdle WL, Mcknight B, Mohlke KL, Mihailov E, Milani L, Mills R, Montasser ME, Morris AP, Müller G, Musk AW, Narisu N, Ong KK, Oostra BA, Osmond C, Palotie A, Pankow JS, Paternoster L, Penninx BW, Pichler I, Pilia MG, Polašek O, Pramstaller PP, Raitakari OT, Rankinen T, Rao DC, Rayner NW, Ribel-Madsen R, Rice TK, Richards M, Ridker PM, Rivadeneira F, Ryan KA, Sanna S, Sarzynski MA, Scholtens S, Scott RA, Sebert S, Southam L, Sparsø TH, Steinthorsdottir V, Stirrups K, Stolk RP, Strauch K, Stringham HM, Swertz MA, Swift AJ, Tönjes A, Tsafantakis E, van der Most PJ, Van Vliet-Ostaptchouk JV, Vandenput L, Vartiainen E, Venturini C, Verweij N, Viikari JS, Vitart V, Vohl MC, Vonk JM, Waeber G, Widén E, Willemsen G, Wilsgaard T, Winkler TW, Wright AF, Yerges-Armstrong LM, Hua Zhao J, Carola Zillikens M, Boomsma DI, Bouchard C, Chambers JC, Chasman DI, Cusi D, Gansevoort RT, Gieger C, Hansen T, Hicks AA, Hu F, Hveem K, Jarvelin MR, Kajantie E, Kooner JS, Kuh D, Kuusisto J, Laakso M, Lakka TA, Lehtimäki T, Metspalu A, Njølstad I, Ohlsson C, Oldehinkel AJ, Palmer LJ, Pedersen O, Perola M, Peters A, Psaty BM, Puolijoki H, Rauramaa R, Rudan I, Salomaa V, Schwarz PE, Shudiner AR, Smit JH, Sørensen TI, Spector TD, Stefansson K, Stumvoll M, Tremblay A, Tuomilehto J, Uitterlinden AG, Uusitupa M, Völker U, Vollenweider P, Wareham NJ, Watkins H, Wilson JF, Zeggini E, Abecasis GR, Boehnke M, Borecki IB, Deloukas P, van Duijn CM, Fox C, Groop LC, Heid IM, Hunter DJ, Kaplan RC, McCarthy MI, North KE, O'Connell JR, Schlessinger D, Thorsteinsdottir U, Strachan DP, Frayling T, Hirschhorn JN, Müller-Nurasyid M, Loos RJ. A principal component meta-analysis on multiple anthropometric traits identifies novel loci for body shape. Nat Commun. 2016;7:13357.
Barban N, Jansen R, de Vlaming R, Vaez A, Mandemakers JJ, Tropf FC, Shen X, Wilson JF, Chasman DI, Nolte IM, Tragante V, van der Laan SW, Perry JR, Kong A; BIOS Consortium, Ahluwalia TS, Albrecht E, Yerges-Armstrong L, Atzmon G, Auro K, Ayers K, Bakshi A, Ben-Avraham D, Berger K, Bergman A, Bertram L, Bielak LF, Bjornsdottir G, Bonder MJ, Broer L, Bui M, Barbieri C, Cavadino A, Chavarro JE, Turman C, Concas MP, Cordell HJ, Davies G, Eibich P, Eriksson N, Esko T, Eriksson J, Falahi F, Felix JF, Fontana MA, Franke L, Gandin I, Gaskins AJ, Gieger C, Gunderson EP, Guo X, Hayward C, He C, Hofer E, Huang H, Joshi PK, Kanoni S, Karlsson R, Kiechl S, Kifley A, Kluttig A, Kraft P, Lagou V, Lecoeur C, Lahti J, Li-Gao R, Lind PA, Liu T, Makalic E, Mamasoula C, Matteson L, Mbarek H, McArdle PF, McMahon G, Meddens SF, Mihailov E, Miller M, Missmer SA, Monnereau C, van der Most PJ, Myhre R, Nalls MA, Nutile T, Kalafati IP, Porcu E, Prokopenko I, Rajan KB, Rich-Edwards J, Rietveld CA, Robino A, Rose LM, Rueedi R, Ryan KA, Saba Y, Schmidt D, Smith JA, Stolk L, Streeten E, Tönjes A, Thorleifsson G, Ulivi S, Wedenoja J, Wellmann J, Willeit P, Yao J, Yengo L, Zhao JH, Zhao W, Zhernakova DV, Amin N, Andrews H, Balkau B, Barzilai N, Bergmann S, Biino G, Bisgaard H, Bønnelykke K, Boomsma DI, Buring JE, Campbell H, Cappellani S, Ciullo M, Cox SR, Cucca F, Toniolo D, Davey-Smith G, Deary IJ, Dedoussis G, Deloukas P, van Duijn CM, de Geus EJ, Eriksson JG, Evans DA, Faul JD, Sala CF, Froguel P, Gasparini P, Girotto G, Grabe HJ, Greiser KH, Groenen PJ, de Haan HG, Haerting J, Harris TB, Heath AC, Heikkilä K, Hofman A, Homuth G, Holliday EG, Hopper J, Hyppönen E, Jacobsson B, Jaddoe VW, Johannesson M, Jugessur A, Kähönen M, Kajantie E, Kardia SL, Keavney B, Kolcic I, Koponen P, Kovacs P, Kronenberg F, Kutalik Z, La Bianca M, Lachance G, Iacono WG, Lai S, Lehtimäki T, Liewald DC; LifeLines Cohort Study, Lindgren CM, Liu Y, Luben R, Lucht M, Luoto R, Magnus P, Magnusson PK, Martin NG, McGue M, McQuillan R, Medland SE, Meisinger C, Mellström D, Metspalu A, Traglia M, Milani L, Mitchell P, Montgomery GW, Mook-Kanamori D, de Mutsert R, Nohr EA, Ohlsson C, Olsen J, Ong KK, Paternoster L, Pattie A, Penninx BW, Perola M, Peyser PA, Pirastu M, Polasek O, Power C, Kaprio J, Raffel LJ, Räikkönen K, Raitakari O, Ridker PM, Ring SM, Roll K, Rudan I, Ruggiero D, Rujescu D, Salomaa V, Schlessinger D, Schmidt H, Schmidt R, Schupf N, Smit J, Sorice R, Spector TD, Starr JM, Stöckl D, Strauch K, Stumvoll M, Swertz MA, Thorsteinsdottir U, Thurik AR, Timpson NJ, Tung JY, Uitterlinden AG, Vaccargiu S, Viikari J, Vitart V, Völzke H, Vollenweider P, Vuckovic D, Waage J, Wagner GG, Wang JJ, Wareham NJ, Weir DR, Willemsen G, Willeit J, Wright AF, Zondervan KT, Stefansson K, Krueger RF, Lee JJ, Benjamin DJ, Cesarini D, Koellinger PD, den Hoed M, Snieder H, Mills MC. Genome-wide analysis identifies 12 loci influencing human reproductive behavior. Nat Genet. 2016;48:1462-72.
Horikoshi M, Beaumont RN, Day FR, Warrington NM, Kooijman MN, Fernandez-Tajes J, Feenstra B, van Zuydam NR, Gaulton KJ, Grarup N, Bradfield JP, Strachan DP, Li-Gao R, Ahluwalia TS, Kreiner E, Rueedi R, Lyytikäinen LP, Cousminer DL, Wu Y, Thiering E, Wang CA, Have CT, Hottenga JJ, Vilor-Tejedor N, Joshi PK, Boh ET, Ntalla I, Pitkänen N, Mahajan A, van Leeuwen EM, Joro R, Lagou V, Nodzenski M, Diver LA, Zondervan KT, Bustamante M, Marques-Vidal P, Mercader JM, Bennett AJ, Rahmioglu N, Nyholt DR, Ma RC, Tam CH, Tam WH; CHARGE Consortium Hematology Working Group, Ganesh SK, van Rooij FJ, Jones SE, Loh PR, Ruth KS, Tuke MA, Tyrrell J, Wood AR, Yaghootkar H, Scholtens DM, Paternoster L, Prokopenko I, Kovacs P, Atalay M, Willems SM, Panoutsopoulou K, Wang X, Carstensen L, Geller F, Schraut KE, Murcia M, van Beijsterveldt CE, Willemsen G, Appel EV, Fonvig CE, Trier C, Tiesler CM, Standl M, Kutalik Z, Bonàs-Guarch S, Hougaard DM, Sánchez F, Torrents D, Waage J, Hollegaard MV, de Haan HG, Rosendaal FR, Medina-Gomez C, Ring SM, Hemani G, McMahon G, Robertson NR, Groves CJ, Langenberg C, Luan J, Scott RA, Zhao JH, Mentch FD, MacKenzie SM, Reynolds RM; Early Growth Genetics (EGG) Consortium, Lowe WL, Tönjes A, Stumvoll M, Lindi V, Lakka TA, van Duijn CM, Kiess W, Körner A, Sørensen TI, Niinikoski H, Pahkala K, Raitakari OT, Zeggini E, Dedoussis GV, Teo YY, Saw SM, Melbye M, Campbell H, Wilson JF, Vrijheid M, de Geus EJ, Boomsma DI, Kadarmideen HN, Holm JC, Hansen T, Sebert S, Hattersley AT, Beilin LJ, Newnham JP, Pennell CE, Heinrich J, Adair LS, Borja JB, Mohlke KL, Eriksson JG, Widén E, Kähönen M, Viikari JS, Lehtimäki T, Vollenweider P, Bønnelykke K, Bisgaard H, Mook-Kanamori DO, Hofman A, Rivadeneira F, Uitterlinden AG, Pisinger C, Pedersen O, Power C, Hyppönen E, Wareham NJ, Hakonarson H, Davies E, Walker BR, Jaddoe VW, Järvelin MR, Grant SF, Vaag AA, Lawlor DA, Frayling TM, Smith GD, Morris AP, Ong KK, Felix JF, Timpson NJ, Perry JR, Evans DM, McCarthy MI, Freathy RM. Genome-wide associations for birth weight and correlations with adult disease. Nature. 2016;538:248-52.
Guiu-Jurado E, Unthan M, Böhler N, Kern M, Landgraf K, Dietrich A, Schleinitz D, Ruschke K, Klöting N, Faßhauer M, Tönjes A, Stumvoll M, Körner A, Kovacs P, Blüher M. Bone morphogenetic protein 2 (BMP2) may contribute to partition of energy storage into visceral and subcutaneous fat depots. Obesity (Silver Spring). 2016;24:2092-100.
Winkler TW, Justice AE, Graff M, [...], Blüher M, [...], Kovacs P, [...], Tönjes A, [...], Stumvoll M, [...], Loos RJ. Correction. The influence of age and sex on genetic associations with adult body size and shape: A large-scale genome-wide interaction study. PLoS Genet. 2016;12:e1006166.
Lazaridis I, Nadel D, Rollefson G, Merrett DC, Rohland N, Mallick S, Fernandes D, Novac M, Gamarra B, Sirak K, Connell S, Stewardson K, Harney E, Fu Q, Gonzales-Fortes G, Jones ER, Roodenberg SA, Lengyel G, Bocquentin F, Gasparian B, Monge JM, Gregg M, Eshed V, Mizrahi AS, Meiklejohn C, Gerritsen F, Bejenaru L, Blüher M, Campbell A, Cavalleri G, Comas D, Froguel P, Gilbert E, Kerr SM, Kovacs P, Krause J, McGettigan D, Merrigan M, Merriwether DA, O'Reilly S, Richards MB, Semino O, Shamoon-Pour M, Stefanescu G, Stumvoll M, Tönjes A, Torroni A, Wilson JF, Yengo L, Hovhannisyan NA, Patterson N, Pinhasi R, Reich D. Genomic insights into the origin of farming in the ancient Near East. Nature. 2016;536:419-24.
Pfeiffer S, Krüger J, Maierhofer A, Böttcher Y, Klöting N, El Hajj N, Schleinitz D, Schön MR, Dietrich A, Fasshauer M, Lohmann T, Dreßler M, Stumvoll M, Haaf T, Blüher M, Kovacs P. Hypoxia-inducible factor 3A gene expression and methylation in adipose tissue is related to adipose tissue dysfunction. Sci Rep. 2016;6:27969.
Grellmann C, Neumann J, Bitzer S, Kovacs P, Tönjes A, Westlye LT, Anreassen OA, Stumvoll M, Villringer A, Horstmann A. Random projection for fast and efficient multivariate correlation analysis of high-dimensional data: A new approach. Front Genet. 2016;7:102.
Prasad RB, Lessmark A, Almgren P, Kovacs G, Hansson O, Oskolkov N, Vitai M, Ladenvall C, Kovacs P, Fadista J, Lachmann M, Zhou Y, Sonestedt E, Poon W, Wollheim CB, Orho-Melander M, Stumvoll M, Tuomi T, Pääbo S, Koranyi L, Groop L. Excess maternal transmission of variants in the THADA gene to offspring with type 2 diabetes. Diabetologia. 2016;59:1702-13.
Voisin S, Almen MS, Zheleznyakova GY, Lundberg L, Zarei S, Castillo S, Eriksson FE, Nilsson EK, Blüher M, Böttcher Y, Kovacs P, Klovins J, Rask-Andersen M, Schiöth HB. Many obesity-associated SNPs strongly associate with DNA methylation changes at proximal promoters and enhancers. Genome Med. 2015;7:103.
Winkler TW, Justice AE, Graff M, [...], Blüher M, [...], Kovacs P, [...], Stumvoll M, [...], Loos RJ. The influence of age and sex on genetic associations with adult body size and shape: A large-scale genome-wide interaction study. PLoS One. 2015;11:e1005378.
Burkhardt R, Kirsten H, Beutner F, Holdt LM, Gross A, Teren A, Tönjes A, Becker S, Krohn K, Kovacs P, Stumvoll M, Teupser D, Thiery J, Ceglarek U, Scholz M. Integration of genome-wide SNP data and gene-expression profiles reveals six novel loci and regulatory mechanisms for amino acids and acylcarnitines in whole blood. PLoS Genet. 2015;11:e1005510.
Schunter JA, Löffler D, Wiesner T, Kovacs P, Badenhoop K, Aust G, Tönjes A, Müller P, Baber R, Simon JC, Führer D, Pfäffle RW, Thiery J, Stumvoll M, Kiess W, Kratzsch J, Körner A. A novel FoxD3 variant is associated with vitiligo and elevated thyroid auto-antibodies. J Clin Endocrinol Metab. 2015;100:E1335-42.
Popkova Y, Meusel A, Breitfeld J, Schleinitz D, Hirrlinger J, Dannenberger D, Kovacs P, Schiller J. Nutrition-dependent changes of mouse adipose tissue compositions monitored by NMR, MS, and chromatographic methods. Anal Bioanal Chem. 2015;407:5113-23.
Rohde K, Federbusch M, Horstmann A, Keller M, Villringer A, Stumvoll M, Tönjes A, Kovacs P, Böttcher Y. Genetic variants in AKR1B10 associate with human eating behavior. BMC Genet. 2015;16:31.
Knigge A, Klöting N, Schön MR, Dietrich A, Fasshauer M, Gärtner D, Lohmann T, Dreßler M, Stumvoll M, Kovacs P, Blüher M. ADCY5 gene expression in adipose tissue is related to obesity in men and mice. PLoS One. 2015;10:e0120742.
Rohde K, Keller M, Horstmann A, Liu X, Eichelmann F, Stumvoll M, Villringer A, Kovacs P, Tönjes A, Böttcher Y. Role of genetic variants in ADIPOQ in human eating behavior. Genes Nutr. 2015;10:449.
Shungin D, Winkler TW, Croteau-Chonka DC, Ferreira T, Locke AE, Mägi R, Strawbridge RJ, Pers TH, Fischer K, [….], Kovacs P, [….], Zillikens MC, McCarthy MI, Speliotes EK, North KE, Fox CS, Barroso I, Franks PW, Ingelsson E, Heid IM, Loos RJ, Cupples LA, Morris AP, Lindgren CM, Mohlke KL. New genetic loci link adipose and insulin biology to body fat distribution. Nature. 2015;518:187-96.
Kern M, Kosacka J, Brückner J, Hesselbarth N, Heiker J, Flehmig G, Klöting I, Kovacs P, Matz-Soja M, Gebhardt R, Krohn K, Sales S, Shevchenko A, Abshagen K, Stumvoll M, Blüher M, Klöting N. Liver-restricted Repin1 Deficiency Improves Whole Body Insulin Sensitivity, Alters Lipid Metabolism and Causes Secondary Changes in Adipose Tissue in Mice. Diabetes. 2014:63:3295-309.
Keller M, Kralisch S, Rohde K, Schleinitz D, Dietrich A, Schön MR, Gärtner D, Lohmann T, Dreßler M, Tönjes A, Stumvoll M, Kovacs P, Faßhauer M, Blüher M, Böttcher Y. Global DNA methylation levels in human adipose tissue are related to fat distribution and glucose homeostasis. Diabetologia. 2014;57:2374-83.
Derikx MH, Kovacs P, Scholz M, Masson E, Chen JM, Ruffert C, Lichtner P, Te Morsche RH, Cavestro GM, Algül H, Berg T, Bödeker H, Blüher M, Bruno MJ, Buch S, Bugert P, Cichoz-Lach H, Dabrowski A, Farré A, Frank J, Gasiorowska A, Geisz A, Goni E, Grothaus J, Grützmann R, Haas S, Hampe J, Hellerbrand C, Hegyi P, Huster D, Ioana M, Iordache S, Jurkowska G, Keim V, Landt O, Di Leo M, Lerch MM, Lévy P, Löhr MJ, Macek M, Malats N, Malecka-Panas E, Mariani A, Martorana D, Mayerle J, Mora J, Mössner J, Müller S, Ockenga J, Paderova J, Pedrazzoli S, Pereira SP, Pfützer R, Real FX, Rebours V, Ridinger M, Rietschel M, Rohde K, Sack S, Saftoiu A, Schneider A, Schulz HU, Soyka M, Simon P, Skipworth J, Stickel F, Stumvoll M, Testonin PA, Tönjes A, Treiber M, Weiss FU, Werner J, Wodarz N, Férec C, Drenth JP, Witt H, Rosendahl. Polymorphisms at PRSS1-PRSS2 and CLDN2-MORC4 loci associate with alcoholic and non-alcoholic chronic pancreatitis in a European replication study. Gut. 2014;64:1426-33.
Tönjes A, Scholz M, Breitfeld J, Marzi C, Grallert H, Gross A, Ladenvall C, Schleinitz D, Krause K, Kirsten H, Laurila E, Kriebel J, Thorand B, Rathmann W, Groop L, Prokopenko I, Isomaa B, Beutner F, Kratzsch J, Thiery J, Fasshauer M, Klöting N, Gieger C, Blüher M, Stumvoll M, Kovacs P. Genome wide meta-analysis highlights the role of genetic variation in RARRES2 in the regulation of circulating serum chemerin. PLoS Genet. 2014;10:e1004854.
Rohde K, Keller M, Klös M, Schleinitz D, Dietrich A, Schön MR, Gärtner D, Lohmann T, Dreßler M, Stumvoll M, Kovacs P, Blüher M, Böttcher Y. Adipose tissue depot specific promotor methylation of TMEM18. J Mol Med. 2014;92:881-8.
Schleinitz D, Böttcher Y, Blüher M, Kovacs P. The genetics of fat distribution. Diabetologia. 2014;57:1276-86.
Schleinitz D, Klöting N, Lindgren CM, Breitfeld J, Dietrich A, Schön MR, Lohmann T, Dreßler M, Stumvoll M, McCarthy MI, Blüher M, Kovacs P. Fat depot-specific mRNA expression of novel loci associated with waist-hip ratio. Int J Obes (Lond). 2014;38:120-5.
Keller M, Liu X, Wohland T, Rohde K, Gast MT, Stumvoll M, Kovacs P, Tönjes A, Böttcher Y. TAS2R38 and its influence on smoking behavior and glucose homeostasis in the German Sorbs. PLoS One. 2013;8:e80512.
Hoffmeister A, Tuennemann J, Sommerer I, Mössner J, Rittger A, Schleinitz D, Kratzsch J, Rosendahl J, Klöting N, Stahl T, Rossner S, Paroni F, Maedler K, Kovacs P, Blüher M. Genetic and biochemical evidence for a functional role of BACE1 in the regulation of insulin mRNA expression. Obesity (Silver Spring). 2013;21:E626-33.
Keller M, Schleinitz D, Förster J, Tönjes A, Böttcher Y, Fischer-Rosinsky A, Breitfeld J, Weidle K, Rayner NW, Burkhardt R, Enigk B, Müller I, Halbritter J, Koriath M, Pfeiffer A, Krohn K, Groop L, Spranger J, Stumvoll M, Kovacs P. THOC5 : a novel gene involved in HDL-cholesterol metabolism. J Lipid Res. 2013;54:3170-6.
Heiker JT, Kern M, Kosacka J, Flehmig G, Stumvoll M, Shang E, Lohmann T, Dreßler M, Kovacs P, Blüher M, Klöting N. Nicotinamide nucleotide transhydrogenase mRNA expression is related to human obesity. Obesity (Silver Spring). 2013;21:529-34.
Kovacs P, Miehle K, Sandner B, Stumvoll M, Blüher M. Insulin administration acutely decreases vaspin serum concentrations in humans. Obes Facts. 2013;6:86-8.
Mai M, Tönjes A, Kovacs P, Stumvoll M, Fiedler GM, Leichtle AB. Serum levels of acylcarnitines are altered in prediabetic conditions. PLoS ONE. 2013;8:e82459.
Gast MT, Tönjes A, Keller M, Horstmann A, Steinle N, Scholz M, Müller I, Villringer A, Stumvoll M, Kovacs P, Böttcher Y. The role of rs2237781 within GRM8 in eating behavior. Brain Behav. 2013;5:495-502.
Horstmann A, Kovacs P, Kabisch S, Boettcher Y, Schloegl H, Toenjes A, Stumvoll M, Pleger B, Villringer A. Common genetic variation near MC4R has a sex-specific impact on human brain structure and brain behavior. PLoS One. 2013;8:e74362.
Schleinitz D, Klöting N, Böttcher Y, Wolf S, Dietrich K, Tönjes A, Breitfeld J, Enigk B, Halbritter J, Körner A, Schön MR, Jenkner J, Tseng Y-H, Stumvoll M, Blüher M, Kovacs P. Human Bone Morphogenetic Protein Receptor 2 (BMPR2) is Involved in the Pathophysiology of Obesity. PLoS One. 2011;6:e16155.
Klöting N*, Kovacs P*, Kern M, Heiker JT, Schön MR, Stumvoll M, Beck-Sickinger AG, Blüher M. Central vaspin administration acutely reduces food intake with sustained blood glucose lowering effects. Diabetologia. 2011;54:1819-23.
Schleinitz D, Klöting N, Körner A, Berndt J, Reichenbächer M, Tönjes A, Ruschke K, Böttcher Y, Dietrich K, Enigk B, Filz M, Schön MR, Jenkner J, Kiess W, Stumvoll M, Blüher M, Kovacs P. Effect of genetic variation in the human fatty acid synthase gene (FASN) on obesity and fat depot-specific mRNA expression. Obesity. 2010;18:1218-25.
Heid IM, Jackson AU, Randall JC, Winkler TW, [...], Kovacs P, Kroemer HK, [...], McCarthy MI, Fox CS, Mohlke KL, Lindgren CM. Meta-analysis identifies 13 novel loci associated with waist-hip ratio and reveals sexual dimorphism in the genetic basis of fat distribution. Nat Genet. 2010;42:949-60.
Böttcher Y*, Unbehauen H*, Klöting N, Ruschke K, Körner A, Schleinitz D, Tönjes A, Enigk B, Dietrich K, Koriath M, Scholz GH, Tseng Y, Schön MR, Kiess W, Stumvoll M, Blüher M, Kovacs P. Adipose Tissue Expression and Genetic Variants of the Bone Morphogenetic Protein Receptor 1A Gene (BMPR1A) are Associated with Human Obesity. Diabetes. 2009;58:2119-28.
Klöting N*, Schleinitz D*, Ruschke K, Berndt J, Fasshauer M, Tönjes A, Schön MR, Kovacs P, Stumvoll M, Blüher M. Inverse relationship between obesity and FTO gene expression in visceral adipose tissue in humans. Diabetologia. 2008;51:641-7.
Kovacs P*, Geyer M*, Berndt J, Klöting N, Graham TE, Böttcher Y, Enigk B, Tönjes A, Schleinitz D, Schön MR, Kahn BB, Blüher M, Stumvoll M. Effects of Genetic Variation in the Human Retinol Binding Protein-4 Gene (RBP4) on Insulin Resistance and Fat Depot Specific mRNA Expression. Diabetes. 2007;56:3095-100.
Berndt J, Kralisch S, Kovacs P, Fasshauer M, Schon MR, Stumvoll M, Blüher M. Vaspin gene expression in human adipose tissue: Association with obesity and type 2 diabetes. Biochem Biophys Res Commun. 2006;339:430-6.
(* contributed equally)

Doktorandin
Luise Müller
|
Büroadresse:
|
ZFG, Liebigstraße 19, Room 4006 |
|
Telefon:
E-mail
|
0341 97 16654
|
|
Projekt-
beschriebung:
|
tba
|
Doktorandin
Samantha Aurich
|
Büroadresse:
|
ZFG, Liebigstraße 9, Room 4006 |
|
Telefon:
E-mail:
|
0341 97 16654
|
|
Projektbeschreibung:
|
In meiner Doktorarbeit beschäftige ich mich mit zellfreier, zirkulierender DNA aus Blutproben um gewebespezifische DNA-Methylierungsstrukturen und epigenetische (Neu-)Programmierungen zu detektieren. Diese können dann dabei helfen Patienten mit stoffwechselgesunder von denen mit ungesunder Adipositas zu unterscheiden. Außerdem möchte ich Unterschiede in cfDNA-Methylierungsmustern identifizieren, welche die individuelle Diabetes-remission als Konsequenz des Gewichtsverlustes nach einer metabolischen Operation charakterisieren.
|


Doktorandin
Reena Prajapati
|
Büroadresse:
|
Herzzentrum Strümpellstr. 39, D-04289 Leipzig |
|
Telefon:
E-mail
|
0341 865 1428
|
|
Projekt- beschreibung:
|
Ich interessiere mich für die Charakterisierung der Entzündungsreaktion von Endothelzellen als Reaktion auf molekulare Muster im Zusammenhang mit kardiogenem Schock. Das Projekt konzentriert sich auf die Auswirkungen von zirkulierenden Biomarkern im Zusammenhang mit dem kardiogenen Schock auf mehrere zelluläre Komponenten und zelluläre Systeme. Es wird vorgeschlagen, die Integration aller vier CLIP-Parameter unter hypoxischen Bedingungen durchzuführen. Darüber hinaus wird eine sorgfältige Untersuchung durchgeführt, um die regulatorischen miRNAs zu entschlüsseln, die von IL-6 und anderen CLIP-Markern in der Kohorte der Patienten mit kardiogenem Schock beeinflusst werden. Die Validierung dieser Ergebnisse soll durch die Anwendung von Bioinformatik-Tools erfolgen.
|