A2 - Evolutionäre Aspekte von Stoffwechsel- und Adipositas-assoziierten Genen in Primaten
Dieses Projekt wurde von 2013 bis 2016 im Rahmen des SFB 1052 von der Deutschen Forschungsgemeinschaft gefördert.
Die Zunahme von Erkrankungen, die mit dem Energie-Metabolismus zusammenhängen, wie Adipositas, Diabetes und koronare Herzkrankheit, kann mit Veränderungen im Lebensstil, die sich in jüngerer Zeit vollzogen haben, in Verbindung stehen. Es ist die Hypothese aufgestellt worden, dass einige der genetischen Ursachen für solche Erkrankungen ihren Ursprung darin haben, dass sich Menschen in der Vergangenheit an eine zur heutigen Umwelt unterschiedliche Umgebung angepasst haben. Hypothesen zum Ursprung einiger dieser Zivilisationskrankheiten, wie zum Beispiel Diabetes und Adipositas, legen nahe, dass früher vorteilhafte Phänotypen heute in einer modernen Umwelt zu erhöhter Anfälligkeit für diese Erkrankungen führen. Hierbei handelt es sich um die sogenannte "Thrifty Gene"-Hypothese. Untersuchungen zu kürzlich stattgefundener positiver Selektion und eine anschließende systematische Analyse der identifizierten genomischen Regionen können daher Erkenntnisse zur genetischen Basis dieser Störungen liefern.
Auch vergleichende Genomsequenzanalysen können benutzt werden, um Positionen zu identifizieren, an denen nicht-menschliche Primaten bekannte menschliche Mendel'sche Krankheitsvarianten tragen. Wenn die Krankheitsvariante in den Primaten nicht das Krankheitsbild hervorruft, dann können entweder Unterschiede in Umwelt oder Lebenserwartung den Unterschied erklären, oder das Genom der nicht-menschlichen Primaten trägt eine kompensatorische Variante, die das Auftreten der Kranheit verhindert. Falls der Unterschied genetisch erklärbar ist, muss der Mensch die kompensatorische Variante verloren haben. In einigen Fällen kann dieser spezifische Verlust der kompensatorischen Variante durch positive Selektion erklärt werden.
Durch die Untersuchung der evolutionären Geschichte der modernen Menschen mit genomischen Methoden ist es möglich, Positionen im Genom zu identifizieren, die sich seit der Trennung von unseren noch lebenden sowie ausgestorbenen Verwandten verändert haben, und Regionen zu lokalisieren, die in der jüngeren menschlichen Geschichte unter Selektion standen. Die vor kurzem erfolgte Sequenzierung mehrerer Primaten-Genome gibt uns die Möglichkeit, diejenigen genetischen Veränderungen zu untersuchen, die erst seit der Trennung des Menschen vom gemeinsamen Vorfahren mit Schimpansen und Bonobos entstanden sind. Die Genome unserer ausgestorbenen homininen Verwandten, der Neandertaler und Denisovaner, liefern uns Erkenntnisse hinsichtlich der jüngsten Veränderungen, die für die Entwicklung von solchen Phänotypen, die wir für für moderne Menschen spezifisch halten, wichtig gewesen sein können. Basierend auf einem Vergleich mit der Genomsequenz der Neandertaler haben wir Regionen adaptiver Evolution (positiver Selektion) im menschlichen Genom ermittelt. Diese Regionen beinhalten einen erhöhten Anteil von Genen, deren Funktion mit dem Metabolismus zusammenhängt. Durch den Vergleich heutiger und ausgestorbener Primatengenome werden wir Gen-Kandidaten für metabolische Störungen identifizieren und priorisieren. Für diese Kandidaten werden wir Variationsdaten in Populationen sammeln und analysieren. Durch die Integration von genetischen und epigenetischen Daten werden wir die evolutionäre Geschichte dieser krankheitsbezogenen Varianten verfolgen und anderen Konsortium-Mitgliedern Ranglisten von Gen- und SNP-Kandidaten zur weiteren funktionalen Analyse zur Verfügung stellen.
Abbildung 1: Vergleichende evolutionäre Genomstudien geben Informationen über die jüngere Selektion im modernen Menschen.
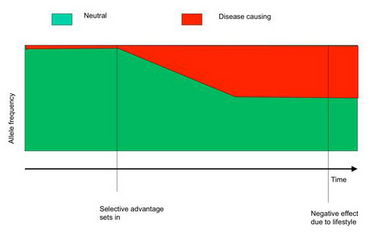
Abbildung 2: Allelfrequenzen in modernen Populationen bieten eine genetische Signatur der Neigung zu Adipositas.
Quach H, Rotival M, Pothlichet J, Loh YE, Dannemann M, Zidane N, Laval G, Patin E, Harmant C, Lopez M, Deschamps M, Naffakh N, Duffy D, Coen A, Leroux-Roels G, Clément F, Boland A, Deleuze JF, Kelso J, Albert ML, Quintana-Murci L. Genetic Adaptation and Neandertal Admixture Shaped the Immune System of Human Populations. Cell. 2016;167:643-656.
Welker F, Hajdinjak M, Talamo S, Jaouen K, Dannemann M, David F, Julien M, Meyer M, Kelso J, Barnes I, Brace S, Kamminga P, Fischer R, Kessler BM, Stewart JR, Pääbo S, Collins MJ, Hublin JJ. Palaeoproteomic evidence identifies archaic hominins associated with the Chatelperronian at the Grotte du Renne. Proc Natl Acad Sci U S A. 2016;113:11162-7.
Fu Q, Posth C, Hajdinjak M, Petr M, Mallick S, Fernandes D, Furtwängler A, Haak W, Meyer M, Mittnik A, Nickel B, Peltzer A, Rohland N, Slon V, Talamo S, Lazaridis I, Lipson M, Mathieson I, Schiffels S, Skoglund P, Derevianko AP, Drozdov N, Slavinsky V, Tsybankov A, Cremonesi RG, Mallegni F, Gely B, Vacca E, Morales MR, Straus LG, Neugebauer-Maresch C, Teschler-Nicola M, Constantin S, Moldovan OT, Benazzi S, Peresani M, Coppola D, Lari M, Ricci S, Ronchitelli A, Valentin F, Thevenet C, Wehrberger K, Grigorescu D, Rougier H, Crevecoeur I, Flas D, Semal P, Mannino MA, Cupillard C, Bocherens H, Conard NJ, Harvati K, Moiseyev V, Drucker DG, Svoboda J, Richards MP, Caramelli D, Pinhasi R,Kelso J, Patterson N, Krause J, Pääbo S, Reich D. The genetic history of Ice Age Europe. Nature. 2016;534:200-5.
Carmody RN, Dannemann M, Briggs AW, Nickel B, Groopmann EE, Wrangham RW, Kelso J. Genetic evidence of human adaption to a cooked diet. Genome Biol Evol. 2016;8:1091-103.
Vernot B, Tucci S, Kelso J, Schraiber JG, Wolf AB, Gittelman RM, Dannemann M, Grote S, McCoy RC, Norton H, Scheinfeldt LB, Merriwether DA, Koki G, Friedlaender JS, Wakefield J, Pääbo S, Akey JM. Excavating Neandertal and Denisovan DNA from the genomes of Melanesian individuals. Science. 2016;352:235-9.
Weiß CL, Dannemann M, Prüfer K, Burbano HA. Contesting the presence of wheat in the British Isles 8,000 years ago by assessing ancient DNA authenticity from low-coverage data. Elife. 2015;4.
Dannemann M, Andres AM, Kelso J. Introgression of Neandertal- and Denisovan-like haplotypes contributesto adaptive variation in human toll-like receptors. Am J Hum Genet. 2016;98:22-33.
Ronke C, Dannemann M, Halbwax M, Fischer A, Helmschrodt C, Brügel M, André C, Atencia R, Mugisha L, Scholz M, Ceglarek U, Thiery J, Pääbo S, Prüfer K, Kelso J. Lineage-specific changes in biomarkers in great apes and humans. PLoS One. 2015;10:e0134548.
Kelso J, Prüfer K. Ancient humans and the origin of modern humans. Curr Opin Gen Dev. 2014;29:133-8.
Khrameeva EE, Bozek K, He L, Yan Z, Jiang X, Wei Y, Tang K, Gelfand MS, Prufer K, Kelso J, Paabo S, Giavalisco P, Lachmann M, Khaitovich P. Neanderthal ancestry drives evolution of lipid catabolism in contemporary Europeans. Nat Commun. 2014;5:3584.
Burbano HA, Green RE, Maricic T, Lalueza-Fox C, de la Rasilla M, Rosas A, Kelso J, Pollard KS, Lachmann M, Paabo S. Analysis of human accelerated DNA regions using archaic hominin genomes. PLoS One. 2012; 7:e32877.
Dannemann M, Nickel B, Lizano E, Burbano HA, Kelso J. Annotation of primate miRNAs by high throughput sequencing of small RNA libraries. BMC Genomics. 2012;13:116.
Dannemann M, Prufer K, Lizano E, Nickel B, Burbano HA, Kelso J. Transcription factors are targeted by differentially expressed miRNAs in primates. Genome Biol Evol. 2012;4:552-64.
Green RE, Krause J, Briggs AW, Maricic T, Stenzel U, [...], Kelso J, Lachmann M, Reich D, Pääbo S. A draft sequence of the Neandertal genome. Science. 2010; 328:710-22.
Kircher M, Kelso J. High-throughput DNA sequencing-concepts and limitations. Bioessays. 2010; 32: 524.
Reich D, Green RE, [...], Hublin JJ, Kelso J, Slatkin M, Pääbo S. Genetic history of an archaic hominin group from Denisova Cave in Siberia. Nature. 2010; 468: 1044-5.
Kircher M, Stenzel U, Kelso J. Improved base calling for the Illumina Genome Analyzer using machine learning strategies. Genome Biol. 2009;10:R83.
Green RE, Malaspinas AS, Krause J, Briggs AW, Johnson PL, Uhler C, Meyer M, Good JM, Maricic T, Stenzel U, Prufer K, Siebauer M, Burbano HA, Ronan M, Rothberg JM, Egholm M, Rudan P, Brajkovic D, Kucan Z, Gusic I, Wikstrom M, Laakkonen L, Kelso J, Slatkin M, Paabo S. A complete Neandertal mitochondrial genome sequence determined by high-throughput sequencing. Cell. 2008; 134: 416-26.
Khaitovich P*, Kelso J*, Franz H, Visagie J, Giger T, Joerchel S, Petzold E, Green RE, Lachmann M, Paabo S. Functionality of intergenic transcription: an evolutionary comparison. PLoS Genet. 2006; 2: e171. (*Beide Autoren haben gleichgewichtig beigetragen.)
Carninci P, Kasukawa T, Katayama S, Gough J, Frith MC, [...], Kelso J, [...], FANTOM Consortium, RIKEN Genome Exploration Research Group and Genome Science Group (Genome Network Project Core Group. The transcriptional landscape of the mammalian genome. Science. 2005; 309: 1559-63.
Tiffin N, Kelso JF, Powell AR, Pan H, Bajic VB, Hide WA. Integration of text- and data-mining using ontologies successfully selects disease gene candidates. Nucleic Acids Res. 2005; 33: 1544-52.
PROJECT TEAM
Janet Kelso, PhD,
Max-Planck-Institut für evolutionäre Anthropologie
Deutscher Platz 6
04103 Leipzig