Z4 - Bioinformatik Unterstützung für den SFB
In der 3ten Förderperiode werden wir den Gruppen im SFB weiterhin vielfältigen bioinformatischen Support anbieten. Dies umfasst die Beratung, Entwicklung und Ausführung von Omic Analysen, die auch kontinuierlich als Galaxy Workflows implementiert werden. Um die Nutzbarkeit zu garantieren und auf dem Stand der Zeit zu bleiben werden die Galaxy-Instanz selbst, sowie die entwickelten Workflows fortwährend gewartet, aktualisiert und angepasst. Der methodische Schwerpunkt der 3ten Förderperiode wird auf der Integration von multi-omics Daten mittels Netzwerkmethoden liegen.
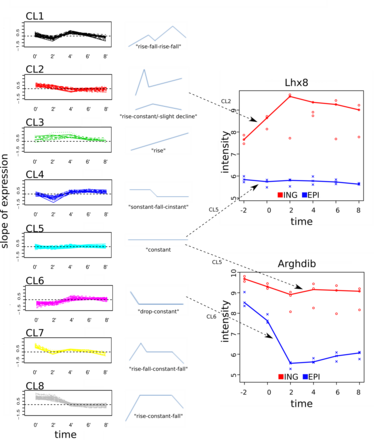
Abbildung 1: Neuartiges Cluster-Verfahren zum Vergleich der Genexpressions-Zeitverläufe in verschiedenen Fettgeweben.
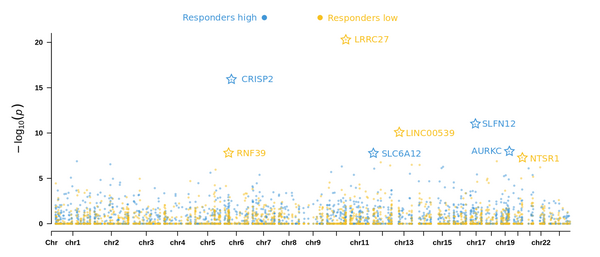
Abbildung 2: Epigenomischer Associationsstudie zu Gewichtsverlust konnte Gene identifizieren, deren Methylierungsrate signifikant mit hoch Respondern (blau) bzw niedrig Respondern (gelb) korrelieren.
Krieg L, Didt K, Karkossa I, Bernhart SH, Kehr S, Subramanian N, Lindhorst A, Schaudinn A, Tabei S, Keller M, Stumvoll M, Dietrich A, von Bergen M, Stadler PF, Laurencikiene J, Krüger M, Blüher M, Gericke M, Schubert K, Kovacs P, Chakaroun R, Massier L. Gut. 2021 Oct 1:gutjnl-2021-324603.
Kolora SRR, Gysi DM, Schaffer S, Grimm-Seyfarth A, Szabolcs M, Faria R, Henle K, Stadler PF, Schlegel M, Nowick K. Accelerated evolution of tissue-specific genes mediates divergence amidst gene flow in European green lizards. Genome Biol Evol. 2021 May 14:evab109.
Yaskolka Meir A, Keller M, Müller L, Bernhart SH, Tsaban G, Zelicha H, Rinott E, Kaplan A, Gepner Y, Shelef I, Schwarzfuchs D, Ceglarek U, Stadler P, Blüher M, Stumvoll M, Kovacs P, Shai I. Effects of lifestyle interventions on epigenetic signatures of liver fat: CENTRAL randomized controlled trial. Liver Int. 2021 May 3.
Meir A Y, Keller Maria, Bernhart S H, Rinott E, Tsaban G, Zelicha H, Kaplan A, Schwarzfuchs D, Shelef I, Gepner Y, Li J, Lin Y, Blüher M, Ceglarek U, Stumvoll M, Stadler P F, Stampfer M J, Kovacs P, Liang L, Shai I. Lifestyle weight‑loss intervention may attenuate methylation aging: the CENTRAL MRI randomized controlled trial. Clin Epigenetics. 2021 Mar 4;13(1):48.
Lorenz R, Stadler PF. RNA Secondary Structures with Limited Base Pair Span: Exact Backtracking and an Application. Genes (Basel). 2020 Dec 24;12(1):E14.
Hoser SM, Hoffmann A, Meindl A, Gamper M, Fallmann J, Bernhart SH, Müller L, Ploner M, Misslinger M, Kremser L, Lindner H, Geley S, Schaal H, Stadler PF, Huettenhofer A. Intronic tRNAs of mitochondrial origin regulate constitutive and alternative splicing. Genome Biol. 2020 Dec 8;21(1):299.
Keller M, Yaskolka Meir A, Bernhart SH, Gepner Y, Shelef I, Schwarzfuchs D, Tsaban G, Zelicha H, Hopp L, Müller L, Rohde K, Böttcher Y, Stadler PF, Stumvoll M, Blüher M, Kovacs P, Shai I. DNA methylation signature in blood mirrors successful weight-loss during lifestyle interventions: the CENTRAL trial. Genome Med. 2020 Nov 16;12(1):97.
Nono Nankam PA, Blüher M, Kehr S, Klöting N, Krohn K, Adams K, Stadler PF, Mendham AE, Goedecke JH. Distinct abdominal and gluteal adipose tissue transcriptome signatures are altered by exercise training in African women with obesity. Sci Rep. 2020 Jun 24;10(1):10240.
Westermann AJ, Förstner KU, Amman F, Barquist L, Chao Y, Schulte LN, Müller L, Reinhardt R, Stadler PF, Vogel J. Dual RNA-seq unveils noncoding RNA functions in host-pathogen interactions. Nature. 2016;529:496-501.
Jühling F, Kretzmer H, Bernhart SH, Otto C, Stadler PF, Hoffmann S. metilene: fast and sensitive calling of differentially methylated regions from bisulfite sequencing data. Genome Res. 2016;6:256-62.
Liu X, Hinney A, Scholz M, Scherag A, Tönjes A, Stumvoll M, Stadler PF, Hebebrand J, Böttcher Y. Indications for potential parent-of-origin effects within the FTO gene. PLoS One. 2015;10:e0119206.
Hoffmann S, Stadler PF, Strimmer K. A simple data-adaptive probabilistic variant calling model. Algorithms Mol Biol. 2015;10:10.
Hoffmann S, Otto C, Doose G, Tanzer A, Langenberger D, Christ S, Kunz M, Holdt LM, Teupser D, Hackermüller J, Stadler PF. A multi-split mapping algorithm for circular RNA, splicing, trans-splicing and fusion detection. Genome Biol. 2014;15:R34.
Otto C, Stadler PF, Hoffmann S. Lacking alignments? The next-generation sequencing mapper segemehl revisited. Bioinformatics. 2014;30:1837-43.
Hackermüller J, Reiche K, Otto C, Hösler N, Blumert C, Brocke-Heidrich K, Böhlig L, Nitsche A, Kasack K, Ahnert P, Krupp W, Engeland K, Stadler PF, Horn F. Cell cycle, oncogenic and tumor suppressor pathways regulate numerous long and macro non-protein-coding RNAs. Genome Biol. 2014;5:R48.
Nitsche A, Doose G, Tafer H, Robinson M, Saha NR, Gerdol M, Canapa A, Hoffmann S, Amemiya CT, Stadler PF. Atypical RNAs in the coelacanth transcriptome. J Exp Zool B Mol Dev Evol. 2014;322:342-51.
Holdt LM, Hoffmann S, Sass K, Langenberger D, Scholz M, Krohn K, Finstermeier K, Stahringer A, Wilfert W, Beutner F, Gielen S, Schuler G, Gäbel G, Bergert H, Bechmann I, Stadler PF, Thiery J, Teupser D. Alu elements in ANRIL non-coding RNA at chromosome 9p21 modulate atherogenic cell functions through trans-regulation of gene networks. PLoS Genet. 2013;9:e1003588.
Otto C, Stadler PF, Hoffmann S. Fast and sensitive mapping of bisulfite-treated sequencing data. Bioinformatics. 2012;28:1698-704.